2019年12月12日,我校生命科学与技术学院陈佳教授、免疫化学研究所杨贝副研究员与中国科学院-马普计算生物学伙伴研究所杨力研究员(我校特聘教授),应邀在国际顶尖学术期刊《细胞》(Cell)上发表题为“One Prime for All Editing”的专评论文(Preview),对哈佛大学David Liu教授近期在Nature杂志发表的《Search-and-replace genome editing without double-strand breaks or donor DNA》(无需双链断裂或者供体DNA的“查找并替换”基因组编辑技术)研究进行介绍,详细解读新型基因编辑系统Prime Editing(导向编辑)的工作原理,并展望该领域未来发展方向。
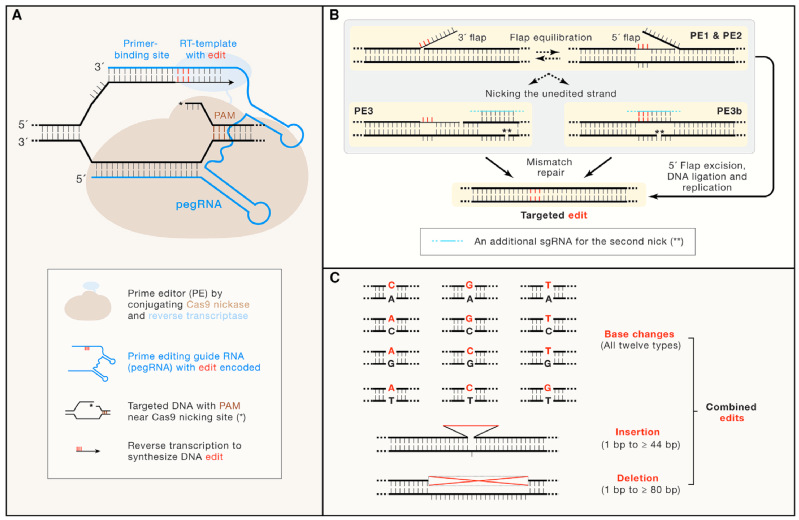
图1:导向编辑器(PE)系统的创建与应用
(A)PE系统示意图(B)PE系统的创建历程(C)PE系统可以介导的编辑类型
在该专评论文中,陈佳教授等对导向编辑系统的设计原理及构建过程进行了介绍(图1)。新型导向编辑系统通过将Cas9切刻酶与逆转录酶融合表达,并利用导向编辑指导RNA(prime editing guide RNA,pegRNA)最终实现靶位点的基因编辑。其中,pegRNA由3个部分组成,包括single-guide RNA(sgRNA)、引物结合位点(Prime Binding Site,PBS)和储存有靶向位点编辑信息的反转录模板(RT templet with edit,图1A)。导向编辑器(Prime Editor,PE,)在创建过程中经历了3步关键改进。PE1利用Cas9切刻酶(H840A)和Moloney Murine Leukemia Virus(M-MLV)逆转录酶构成,虽可以精确实现设计的基因组编辑,但在哺乳动物细胞上的编辑效率较低。因此,研究人员构建了PE2,主要是通过在M-MLV逆转录酶中引入5个氨基酸改变进而提高靶向位点的编辑效率。最后,研究人员构建了PE3/PE3b,通过共表达介导非靶向DNA单链切刻(nick)的sgRNA,利用细胞内源性错配修复(mismatch repair)途径保护编辑链的修饰信息,从而进一步提高了导向编辑的效率(图1B)。导向编辑具有非常广泛的应用前景,可以实现包括12种碱基替换、小片段碱基插入和缺失等的不同编辑用途(图1C),毋庸置疑地将在基础和临床研究领域获得广泛地应用。在这一专评论文中,陈佳教授等也指出了导向编辑系统及其应用仍有亟待改进之处:如gRNA依赖性或非依赖性的脱靶效应尚且未知;PE3介导的高碱基插入/缺失率以及导向编辑系统在成体动物中的递送等问题。
陈佳教授、杨贝副研究员与杨力研究员为该论文的共同通讯作者。
文章链接:https://www.cell.com/cell/fulltext/S0092-8674(19)31287-5


