我校生命科学与技术学院陈佳课题组、黄行许课题组与中国科学院-马普计算生物学研究所研究员杨力(我校生命学院特聘教授)研究组开展合作研究,成功开发出一系列基于人胞嘧啶脱氨酶APOBEC的新型普适碱基编辑器,其中基于人APOBEC3A(hA3A)的碱基编辑器可高效介导甲基化胞嘧啶mC到胸腺嘧啶T的编辑。北京时间8月20日,相关工作以“Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion”为题,在知名学术期刊《自然-生物技术》(Nature Biotechnology)上在线发表。
近年来,将CRISPR/Cas 基因编辑酶(如CRISPR/Cas9、CRISPR/Cpf1等)与核酸脱氨编辑酶(如胞嘧啶脱氨酶APOBEC/AID、腺嘌呤脱氨酶ADAR等)整合发展出的碱基编辑系统(Base Editor,BE),可在单碱基水平对基因实现高效率的靶向编辑改造。这种新型碱基编辑系统理论上可对数百种引起人类疾病的基因组单碱基突变进行定点矫正,因此拥有巨大的临床应用潜力。碱基编辑技术于2017年被《科学》杂志评为全球十大年度科学突破之一,进一步凸显出该领域在科学研究和临床应用上的重要潜力。
在这项最新的研究中,合作团队首先利用生物信息学方法系统分析了与人类疾病相关的单碱基突变,发现很多胸腺嘧啶T到胞嘧啶C的突变大部分处于CpG二核苷酸位点。而在哺乳动物基因组中,CpG位点的胞嘧啶通常易被甲基化修饰。目前已报导的碱基编辑系统大多是利用大鼠APOBEC1(rA1)作为脱氨酶进行基因组编辑,合作团队的研究发现此类rA1依赖的碱基编辑通常会受到DNA甲基化修饰水平的影响,因此在基因组DNA高甲基化区域无法实现高效的碱基编辑。
为了实现高甲基化区域内的高效碱基编辑,合作团队利用十余种来自不同物种的APOBEC胞嘧啶脱氨酶家族蛋白构建出一系列新型碱基编辑器,可广泛用于胞嘧啶C至胸腺嘧啶T单碱基编辑。更为重要的是,合作团队通过一系列筛选分析,发现基于人APOBEC3A的碱基编辑器(hA3A-BE)可在基因组高甲基化区域实现高效的甲基化胞嘧啶mC至胸腺嘧啶T单碱基编辑。深入研究发现,hA3A-BE是一种普适且高效的碱基编辑器,可在已检测的多种环境中实现胞嘧啶C(或甲基化胞嘧啶mC)至胸腺嘧啶T的高效编辑。最后,通过对人APOBEC3A的系统性改造,研究团队缩小了hA3A-BE的编辑区间,进一步提高了其碱基编辑的精度。与之前报道的基于大鼠APOBEC1的碱基编辑器相比,基于人APOBEC3A的新型碱基编辑器应用范围更加广泛,这为碱基编辑系统在基础研究及未来临床领域的全面深入应用提供了新工具、新方法和新思路。
陈佳课题组长期从事DNA损伤修复机制以及基因编辑相关的研究工作,此前已阐明APOBEC胞嘧啶脱氨酶在CRISPR/Cas9介导的基因编辑过程中产生突变的分子机制(Lei et al., 2018, Nature Structural & Molecular Biology);成功创建增强型Cas9碱基编辑器(Wang et al., 2017, Cell Research)以及可在基因组A/T富集区域内开展有效编辑的Cpf1碱基编辑器(Li et al., 2018, Nature Biotechnology);陈佳教授还应邀接受《细胞》杂志采访探讨基因编辑领域的未来发展方向(Chen et al., 2018,Cell)。
该论文中,陈佳课题组2016级硕博连读研究生王潇、黄行许课题组2015级硕博连读研究生李佳楠、免疫化学研究所杨贝博士和中科院计算生物学研究所杨力研究组2015级硕博连读研究生王滢和研究助理韦佳为共同第一作者,陈佳、杨力、黄行许为共同通讯作者,上科大为第一完成单位。该项研究得到了国家自然科学基金委、科技部、上海市科委和上科大科研启动基金的支持。
文章链接:https://www.nature.com/articles/nbt.4198
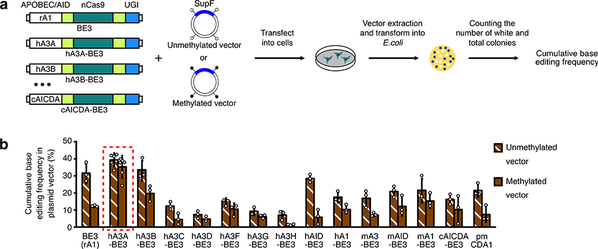
a:构建多种碱基编辑器并筛选其在高甲基化区域的编辑效率
b:hA3A-BE3能够在高甲基化区域进行高效碱基编辑


