近日,上海科技大学生命学院马涵慧课题组在学术期刊 Protein & Cell 上发表研究论文,报道了三种高效灵活的先导基因编辑工具的开发:3’-Stem loop PEs(sPEs)、Tethered PEs(tPEs)和Split pegRNA PEs(SnPEs)。
绝大多数遗传性疾病都是由于基因上的位点突变、插入或缺失导致的。2019年,MIT的David Liu团队开发出了一款基于CRISPR的先导编辑(Prime editor,PE)编辑器,这款编辑器能够在不引入双链DNA断裂的情况下,无需额外添加供体模板,便可实现点突变、DNA片段插入或筛除。但是PE编辑器在很多基因位点的编辑效率非常低,因此,提高PE的编辑效率仍是一个亟需解决的问题。
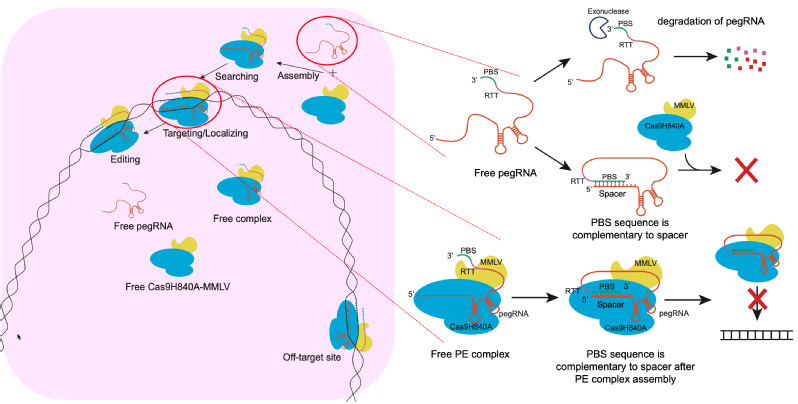
图1 | PE编辑器发挥作用需要经过以下五个步骤:组装、搜索、靶向、定位、编辑。在这个过程中,pegRNA被降解或者自身环化以及pegRNA的3’端在复合物形成后进入Cas9与5’端的spacer结合都会影响对目标位点的靶向,最终影响编辑效率。
团队从先导编辑器靶向目标DNA效率入手研究,利用团队此前开发的CRISPRainbow成像技术,发现PE编辑器的DNA靶向效率非常低,这有可能由于pegRNA自身的稳定性或非正常的折叠,也可能是由于组装的Cas9/pegRNA的DNA靶向能力下降。而其目标DNA靶向能力可以通过在pegRNA的3’-末端加RNA适配体(比如pegRNA-MS2)得到恢复 。

图2 | 使用CRISPR成像系统的三种系统的示意图。在使用GE和PE-MS2系统的细胞中可以观察到有亮点出现,说明这两种系统可以很好地靶向目标位点;在使用PE系统的细胞中几乎无法观察到有亮点出现,说明PE系统不能很好地靶向目标位点。
基于这一发现,团队开发了一系列优化的PE基因编辑系统,包括3’-Stem loop PEs(sPEs)、Tethered PEs(tPEs)和Split pegRNA PEs(SnPEs)。通过在pegRNA的3’端添加RNA 适配体(sPE)以及在Cas9上连接RNA 适配体的结合蛋白(tPE),发现sPE和tPE系统在不同细胞中的编辑效率相比于PE均有所提高,尤其是在片段插入与筛除的应用中编辑效率远高于PE系统。同时为了增加更多改造的灵活性,作者将pegRNA拆分为sgRNA和pRNA (prime RNA)。SnPE不仅解决了在插入与缺失大片段时pegRNA难以稳定存在的问题,还为诱导型先导编辑系统(iPEs)提供了良好的基础。
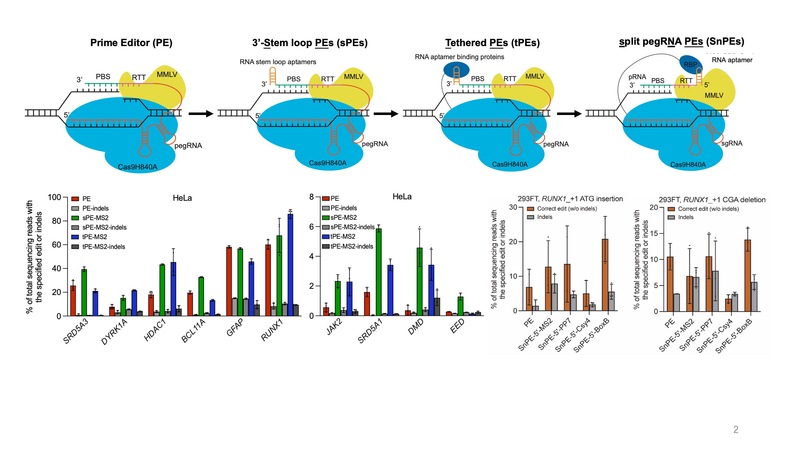
图3 | PE、sPE、tPE、SnPE系统的示意图。PE、sPE和tPE在HeLa细胞中10个不同位点的基因编辑效率。使用不同RNA适配体的SnPE系统在293FT细胞中RUNX1位点诱导三碱基插入和删除的编辑效率。
上科大生命学院访问生、华东理工大学博士研究生冯颖,上海科技大学生命学院2020级研究生刘思远为共同第一作者,上海科技大学生命学院助理教授马涵慧为通讯作者。
论文标题:Enhancing Prime Editing Efficiency and Flexibility with Tethered and Split pegRNAs
论文链接:
https://academic.oup.com/proteincell/advance-article/doi/10.1093/procel/pwac014/6645197


