课题组长
姓名:
杨贝助理教授 研究员 博士生导师, PhD, 助理教授
职务:
所在院所:
生命科学与技术学院、免疫化学研究所
荣誉称号:
东方英才拔尖
教育经历:
-
2000/09—2004/06,武汉大学生物学基地班,学士
-
2004/09—2010/07,中国科学院上海生物化学与细胞生物学研究所,博士
博士后及工作经历:
- 2010/08—2013/06,美国国立健康研究院,博士后
- 2013/07—2015/07,美国加州大学伯克利分校,博士后
- 2015/11—2020/09,上海科技大学免疫化学研究所,副研究员
- 2020/10—至今,上海科技大学生命科学与技术学院、免疫化学研究所,研究员, 助理教授(TENURE-TRACK)
课题组简介
研究内容:
实验室综合运用包括整合结构生物学、生物大分子质谱、基因编辑等在内的前沿技术体系,结合多种体外、体内评估手段,致力于解决具有临床意义的重要生物学问题。现阶段,本实验室主要针对一些系列高风险传染性疾病(如艾滋病、疟疾等)以及遗传性罕见病(如地中海贫血、高血脂症等)开展分子病理机制方向的探索,并致力于针对这些疾病鉴定新型治疗靶点和开发相应的预防新手段(如新型疫苗)与治疗新方法(如基因治疗)。我们鼓励学生进行原创性探索、发展批判性思维,也致力于为各位同学提供自由的学术氛围和了解掌握不同前沿技术的机会。
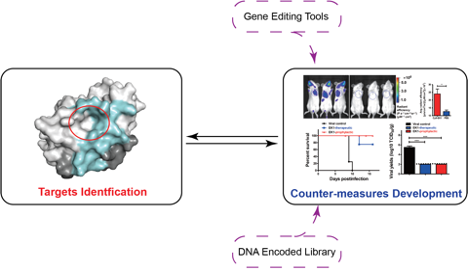
研究成果展示
近5年代表性科研成果:
1、碱基编辑工具开发与应用:通过整合TALE的定位功能和双链DNA胞嘧啶脱氨酶DddA的碱基编辑功能构建而成的线粒体碱基编辑器DdCBEs为线粒体相关遗传性疾病提供了新的治愈可能。然而,DdCBE的脱靶编辑和旁观者编辑限制了它们在临床治疗中的实际应用。为解决上述问题,我们解析了DdCBE分别靶向两个内源性线粒体基因位点的高分辨率冷冻电镜结构,并根据其所揭示的分子机制开发了可用于指导TALE识别区与spacer长度设计的编辑窗口预测模型(WinPred)和高精度型DdCBE(aDdCBE),成功实现了单碱基精度的线粒体DNA编辑和对线粒体疾病的精准模拟,研究成果为未来线粒体疾病的造模和基因治疗提供了有力工具(Mol Cell, 2025)(图1)。在开发和优化基因编辑工具的基础上,我们也初步探索了导向编辑技术在高风险传染性疾病的预防方向的应用潜力(Medcomm, 2023),更与合作者一同推动了新型碱基编辑技术在遗传性疾病–地中海贫血症–治疗中的实际应用,部分临床前概念验证研究成果已发表:该研究利用高精准、无脱靶的变形式碱基编辑系统tBE对γ-珠蛋白的启动子区域进行精准碱基编辑,重新激活了γ-珠蛋白的表达来替代存在基因缺陷的β-珠蛋白,从而重建了血红蛋白的携氧功能(Cell Stem Cell, 2023)。
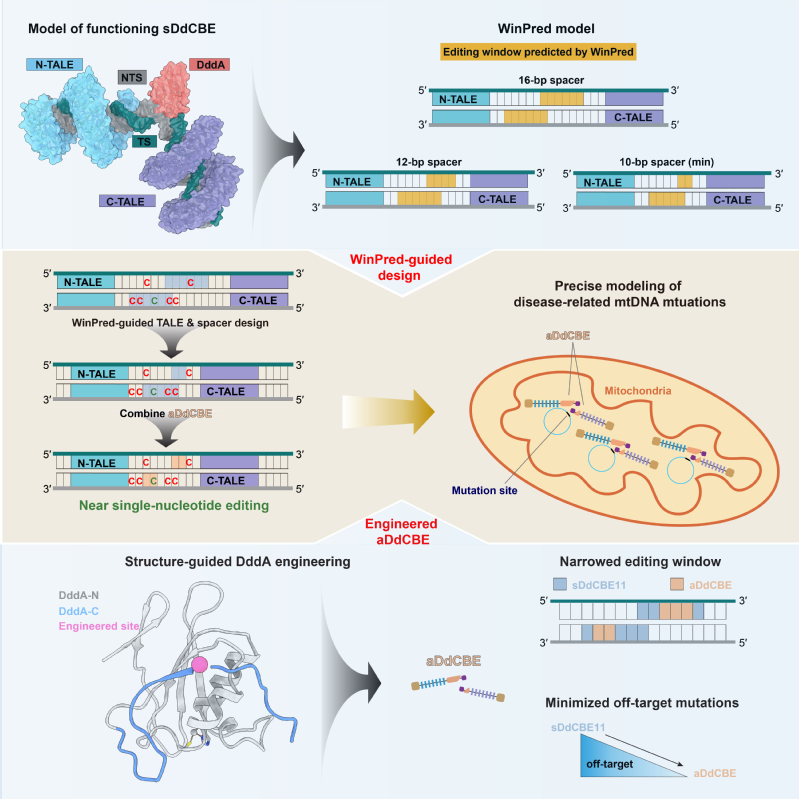
图1、结构指导下的高精准线粒体碱基编辑器开发
2、广谱疫苗靶点发掘:位于HIV-1病毒表面的Env蛋白三聚体是艾滋病疫苗研发的焦点。当前,针对艾滋病疫苗研发困境的破局途径之一是在少数广谱中和抗体的表位指引下开展结构指导的“免疫聚焦型疫苗设计”,其核心思想是通过结构指导的反向蛋白质工程设计,在屏蔽Env蛋白上无效或非广谱表位的同时将广谱中和抗体的抗原表位特异性地呈递给宿主的免疫系统,从而使宿主的免疫应答聚焦在这些保守的“免疫脆弱点”区域,进而引发广谱、高效的体液免疫反应来预防感染。而该策略的开展不仅需要广谱中和抗体抗原表位的指引,更需要对不同亚型Env蛋白的结构与抗原特征进行全面掌握。通过研究,我们报道了我国HIV-1病毒的主流亚型(即流行重组亚型CRF01_AE和CRF07_BC)的Env蛋白结构与免疫识别特征,揭示了CRF01_AE亚型的Env蛋白在V1区域的独有特征(图2A-B)及其与该亚型的广谱中和抗体逃逸能力之间的关系(图2C-D),并阐明了第一株分离自CRF01_AE感染个体的广谱中和抗体的新型中和机制(图2E)。该研究刷新了我们对于HIV-1流行重组亚型的认识,为后续针对我国主流HIV-1亚型开展免疫聚焦型疫苗设计提供了关键信息 (Nat. Commun., 2023)。 此外,我们还描绘了α属冠状病毒刺突蛋白的抗原图谱,揭示了不同属的人冠状病毒的刺突蛋白之间在抗原特性上的共性与差异,相关研究成果也为开发广谱冠状病毒疫苗提供了理论参考(发表于 Commun. Biol., 2022)。
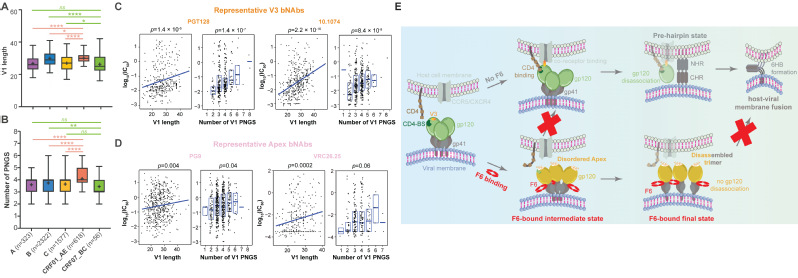
图2、揭示HIV-1流行重组亚型Env蛋白的结构与免疫识别特征。
3、新药靶点发掘与相应抑制剂发现:肠道病毒属(Enterovirus)多种病原体引发的感染因其庞大的感染基数每年给全球社会带来巨大的健康和经济负担,而3C蛋白是针对该属病毒开发抗病毒药物的热点。然而,此前针对其酶活性中心开发的抑制剂在动物实验中效果不佳,亟待发掘3C蛋白上新的药物靶点。我们通过研究发现:1)3C蛋白除具有传统的蛋白酶功能外还在该属病毒的基因组复制过程中发挥了关键调控作用,并鉴定出了3C蛋白上负责与病毒基因组5'NCR区结合的相关位点。2)3C蛋白的酶活性可以通过别构调节位点进行调控。上述别构调节位点和基因组结合位点均远离3C蛋白的酶活性中心,同时具有很高的属内保守性,可用于指导后续针对该属病毒的广谱抑制剂研发工作(发表于PNAS, 2020)。进一步,我们通过综合运用DNA编码化合物库和计算机虚拟筛选等技术针对上述两个新靶点开展了大规模的化合物筛选与验证工作,首次发现天然产物二氢杨梅素(DHM)能特异性靶向3C蛋白的别构调节位点,非竞争性地抑制包括柯萨奇病毒、脊髓灰质炎病毒、肠道病毒71等在内的多种肠道病毒的3C蛋白酶活性,进而通过阻断病毒多蛋白前体的切割来抑制病毒复制,实现广谱抗病毒效应,其在抗病毒实验中的选择性指数CC50/IC50超过700,具备优异的剂量窗口,有望作为抗肠道病毒药物的候选分子被进一步优化和继续开发(Adv Biol, 2025)。
代表性论文(*第一作者,#通讯作者)
- 1. Jiangchao Xiang*, Wenchao Xu*, Jing Wu*, Yaxin Luo, Chengyu Liu, Yaofeng Hou, Jia Chen, Bei Yang.Structural insights into DdCBE in action enable highprecision mitochondrial DNA editing.MOLECULAR CELL. 10 Sep 2025.
- 2. Gao, Yan*; Xie, Xiong; Zhang, Xiaoyu; Cao, Junyuan; Lan, Weiqi; You, Tian; Li, Dongxu; Dong, Xuxue; Dai, Wenhao; Xiang, Yingchun; Hu, Shulei; Shang, Weijuan; Wu, Botao; Zhang, Yumin; Xu, Jin; Liu, Xiaoce; Wang, Haofeng; Hu, Wanlong; Zhang, Mingjing; Duan, Yinkai; Cui, Wen; Zhou, Hao; Mao, Shengjiang; Jia, Handi; Sun, Zhanqi; Jia, Menghan; Yin, Yue; Nguyen, Henry C.; Yang, Kailin; Yang, Bei; Yang, Xiuna; Ji, Xiaoyun; Xiao, Gengfu; Wang, Wei; Zhang, Leike; Rao, Zihe; Liu, Hong; Yang, Haitao.Substrate recognition and cleavage mechanism of the monkeypox virus core protease.NATURE. 01 Apr 2025.
- 3. Chen, Jiayan*; Wang, Qi; He, Xiaomeng; Yang, Bei.Malaria Vaccines: Current Achievements and Path Forward.VACCINES. 19 May 2025. 13(5).
- 4. Sun, Shangwu*; Wang, Qiang; Zhu, Mengyao; Zhang, Xuan; Zhang, Xianfang; Yang, Bei.A Pan-Enterovirus Natural Product Inhibitor Targeting a Unique Allosteric Site on the Viral 3C Protease.ADVANCED BIOLOGY. 01 Jul 2025.
- 5. Guangye Li*; Guo Chen*; Guo-Hua Yuan*; Jia Wei*; Qingyang Ni; Jing Wu; Bei Yang; Li Yang; Jia Chen.Specific and efficient RNA A-to-I editing through cleavage of an ADAR inhibitor.NATURE BIOTECHNOLOGY. 26 Mar 2025.
- 6. Fan, Yuhang*; Xu, Wenchao; Gao, Bao-Qing; Qin, Huichao; Wu, Xiaoyi; Wei, Jia; Ni, Qingyang; Zhou, Lina; Xiang, Jiangchao; Wu, Jing; Yang, Bei; Yang, Li; Chen, Jia.Leveraging base excision repair for efficient adenine base editing of mitochondrial DNA.NATURE BIOTECHNOLOGY. 25 Mar 2025.
- 7. Zhang, Ruiwen*; He, Zhou; Shi, Yajing; Sun, Xiangkun; Chen, Xinyu; Wang, Guoquan; Zhang, Yizhou; Gao, Pan; Wu, Ying; Lu, Shuhan; Duan, Junyi; Sun, Shangwu; Yang, Na; Fan, Wei; Zhao, Kaitao; Yang, Bei; Xia, Yuchen; Zhang, Yan; Zhang, Ying; Yin, Hao.Amplification editing enables efficient and precise duplication of DNA from short sequence to megabase and chromosomal scale.CELL. 25 Jul 2024. 187(15).
- 8. Luo, Yaxin*; Hou, Yaofeng; Zhao, Wenwen; Yang, Bei.Recent progress in gene therapy for familial hypercholesterolemia treatment.ISCIENCE. 20 Sep 2024. 27(9).
- 9. Han, Wenyan*; Qiu, Hou-Yuan*; Sun, Shangwu*; Fu, Zhi-Can*; Wang, Guo-Quan*; Qian, Xiaowen*; Wang, Lijie; Zhai, Xiaowen; Wei, Jia; Wang, Yichuan; Guo, Yi-Lin; Cao, Guo-Hua; Ji, Rui-Jin; Zhang, Yi-Zhou; Ma, Hongxia; Wang, Hongsheng; Zhao, Mingli; Wu, Jing; Bi, Lili; Chen, Qiu-Bing; Li, Zifeng; Yu, Ling; Mou, Xiaodun; Yin, Hao; Yang, Li; Chen, Jia; Yang, Bei; Zhang, Ying.Base editing of the HBG promoter induces potent fetal hemoglobin expression with no detectable off-target mutations in human HSCs.CELL STEM CELL. Nov 2023.
- 10. Niu, Jun*; Wang, Qi; Zhao, Wenwen; Meng, Bing; Xu, Youwei; Zhang, Xianfang; Feng, Yi; Qi, Qilian; Hao, Yanling; Zhang, Xuan; Liu, Ying; Xiang, Jiangchao; Shao, Yiming; Yang, Bei.Structures and immune recognition of Env trimers from two Asia prevalent HIV-1 CRFs.NATURE COMMUNICATIONS. 04 Aug 2023. 14(1).
- 11. Zhao, Wenwen*; Li, Jifang*; Wang, Xiao*; Xu, Wei*; Gao, Bao-Qing*; Xiang, Jiangchao; Hou, Yaofeng; Liu, Wei; Wu, Jing; Qi, Qilian; Wei, Jia; Yang, Xiaoyu; Lu, Lu; Yang, Li; Chen, Jia; Yang, Bei.Prime editor-mediated functional reshaping of ACE2 prevents the entry of multiple human coronaviruses, including SARS-CoV-2 variants.MEDCOMM. 2023.
- 12. Shangwu Sun*; Rui Zhu*; Mengyao Zhu; Qi Wang; Na Li; Bei Yang.Visualization of conformational transition of GRP94 in solution.LIFE SCIENCE ALLIANCE. Nov 2023. 7(2).
- 13. Jiangchao Xiang*; Wenchao Xu*; Jing Wu*; Yaxin Luo; Bei Yang; Jia Chen.Nucleoside deaminases: the key players in base editing toolkit.BIOPHYSICS REPORTS. 01 Nov 2023.
- 14. Jia, Xinshuo*; Li, Yanan*; Wang, Teng; Bi, Lulu; Guo, Lijuan; Chen, Ziting; Zhang, Xia; Ye, Shasha; Chen, Jia; Yang, Bei; Sun, Bo.Discrete RNA–DNA hybrid cleavage by the EXD2 exonuclease pinpoints two rate-limiting steps.THE EMBO JOURNAL. 04 Jan 2023. 42:e111703.
- 15. Xiang, Jiangchao*; Su, Jie; Lan, Qiaoshuai; Zhao, Wenwen; Zhou, Yu; Xu, Youwei; Niu, Jun; Xia, Shuai; Qi, Qilian; Sidhu, Sachdev; Lu, Lu; Miersch, Shane; Yang, Bei.Antigenic mapping reveals sites of vulnerability on alpha-HCoV spike protein.COMMUNICATIONS BIOLOGY. 04 Nov 2022. 5(1).
- 16. Li, Xiaosa*; Zhou, Lina*; Gao, Bao-Qing*; Li, Guangye; Wang, Xiao; Wang, Ying; Wei, Jia; Han, Wenyan; Wang, Zixian; Li, Jifang; Gao, Runze; Zhu, Junjie; Xu, Wenchao; Wu, Jing; Yang, Bei; Sun, Xiaodong; Yang, Li; Chen, Jia.Highly efficient prime editing by introducing same-sense mutations in pegRNA or stabilizing its structure.NATURE COMMUNICATIONS. 29 Mar 2022. 13(1):1669.
- 17. Wang, Lijie*; Xue, Wei*; Zhang, Hongxia*; Gao, Runze*; Qiu, Houyuan*; Wei, Jia; Zhou, Lina; Lei, Yun-Ni; Wu, Xiaocheng; Li, Xiao; Liu, Chengfang; Wu, Jing; Chen, Qiubing; Ma, Hanhui; Huang, Xingxu; Cai, Cheguo; Zhang, Ying; Yang, Bei; Yin, Hao; Yang, Li; Chen, Jia.Eliminating base-editor-induced genome-wide and transcriptome-wide off-target mutations.NATURE CELL BIOLOGY. May 2021. 23(5):552-563.
- 18. Xiaojie Shi*; Yue Wan*; Nan Wang*; Jiangchao Xiang*; Tao Wang; Xiaofeng Yang; Ju Wang; Xuxue Dong; Liang Dong; Lei Yan; Yu Li; Lili Liu; Shinchen Hou; Zhenwei Zhong; Ian A. Wilson; Bei Yang; Guang Yang; Richard A. Lerner.Selection of a picomolar antibody that targets CXCR2-mediated neutrophil activation and alleviates EAE symptoms.NATURE COMMUNICATIONS. 05 May 2021.
- 19. Meng, Bing*; Lan, Keke; Xie, Jia; Lerner, Richard A.; Wilson, Ian A.; Yang, Bei.Inhibitory antibodies identify unique sites of therapeutic vulnerability in rhinovirus and other enteroviruses.PROCEEDINGS OF THE NATIONAL ACADEMY OF SCIENCES OF THE UNITED STATES OF AMERICA. 16 Jun 2020. 117(24):13499-13508.
- 20. Wang, Xiao*; Ding, Chengfeng*; Yu, Wenxia*; Wang, Ying*; He, Siting*; Yang, Bei*; Xiong, Yi-Chun; Wei, Jia; Li, Jifang; Liang, Jiayi; Lu, Zongyang; Zhu, Wei; Wu, Jing; Zhou, Zhi; Huang, Xingxu; Liu, Zhen; Yang, Li; Chen, Jia.Cas12a Base Editors Induce Efficient and Specific Editing with Low DNA Damage Response.CELL REPORTS. 02 Jun 2020. 31(9).
- 21. Cui, Yan-ru*; Wang, Shao-jie*; Chen, Jun; Li, Jie; Chen, Wenzhang; Wang, Shuyue; Meng, Bing; Zhu, Wei; Zhang, Zhuhong; Yang, Bei; Jiang, Biao; Yang, Guang; Ma, Peixiang; Liu, Jia.Allosteric inhibition of CRISPR-Cas9 by bacteriophage-derived peptides.GENOME BIOLOGY. 26 Feb 2020. 21(1).
- 22. Xu, Juncao*; Cui, Kaijie*; Shen, Liqiang; Shi, Jing; Li, Lingting; You, Linlin; Fang, Chengli; Zhao, Guoping; Feng, Yu; Yang, Bei; Zhang, Yu.Crl activates transcription by stabilizing active conformation of the master stress transcription initiation factor.ELIFE. 17 Dec 2019. 8.
- 23. Yang, Li*; Yang, Bei; Chen, Jia.One Prime for All Editing.CELL. Dec 2019. 179(7):1448-1450.
- 24. Zhao, Quanju*; Ren, Chaowei*; Liu, Linyi*; Chen, Jinju; Shao, Yubao; Sun, Ning; Sun, Renhong; Kong, Ying; Ding, Xinyu; Zhang, Xianfang; Xu, Youwei; Yang, Bei; Yin, Qianqian; Yang, Xiaobao; Jiang, Biao.Discovery of SIAIS178 as an Effective BCR-ABL Degrader by Recruiting Von Hippel-Lindau (VHL) E3 Ubiquitin Ligase.JOURNAL OF MEDICINAL CHEMISTRY. 24 Oct 2019. 62(20):9281-9298.
- 25. Wang, Ying*; Gao, Runze*; Wu, Jing*; Xiong, Yi-Chun; Wei, Jia; Zhang, Sipin; Yang, Bei; Chen, Jia; Yang, Li.Comparison of cytosine base editors and development of the BEable-GPS database for targeting pathogenic SNVs.GENOME BIOLOGY. Oct 2019. 20(1).
- 26. Chen, Jia*; Yang, Bei; Yang, Li.To BE or not to BE, that is the question.NATURE BIOTECHNOLOGY. May 2019. 37(5):520-521.
- 27. Xia, Shuai*; Yan, Lei*; Xu, Wei*; Agrawal, Anurodh Shankar; Algaissi, Abdullah; Tseng, Chien-Te K.; Wang, Qian; Du, Lanying; Tan, Wenjie; Wilson, Ian A.; Jiang, Shibo; Yang, Bei; Lu, Lu.A pan-coronavirus fusion inhibitor targeting the HR1 domain of human coronavirus spike.SCIENCE ADVANCES. Apr 2019. 5(4).
- 28. Yang, Bei*; Yang, Li; Chen, Jia.Development and Application of Base Editors.CRISPR JOURNAL. Apr 2019. 2(2):91-104.
- 29. Wang, Xiao*; Li, Jianan*; Wang, Ying*; Yang, Bei*; Wei, Jia*; Wu, Jing; Wang, Ruixuan; Huang, Xingxu; Chen, Jia; Yang, Li.Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion.NATURE BIOTECHNOLOGY. Oct 2018. 36(10):946-949.
- 30. Yan, Lei*; Meng, Bing; Xiang, Jiangchao; Wilson, Ian A.; Yang, Bei.Crystal structure of the post-fusion core of the Human coronavirus 229E spike protein at 1.86 angstrom resolution.ACTA CRYSTALLOGRAPHICA SECTION D-STRUCTURAL BIOLOGY. Sep 2018. 74:841-851.
- 31. Qiang, Min*; Dong, Xue*; Zha, Zhao; Zuo, Xiao-Kun; Song, Xing-Lei; Zhao, Lixia; Yuan, Chao; Huang, Chen; Tao, Pingdong; Hu, Qin; Li, Wei-Guang; Hu, Wanhui; Li, Jie; Nie, Yan; Buratto, Damiano; Zonta, Francesco; Ma, Peixiang; Yu, Zheng; Liu, Lili; Zhang, Yi; Yang, Bei; Xie, Jia; Xu, Tian-Le; Qu, Zhihu; Yang, Guang; Lerner, Richard A..Selection of an ASIC1a-blocking combinatorial antibody that protects cells from ischemic death.PROCEEDINGS OF THE NATIONAL ACADEMY OF SCIENCES OF THE UNITED STATES OF AMERICA. 07 Aug 2018. 115(32):E7469-E7477.
- 32. Li, Xiaosa*; Wang, Ying*; Liu, Yajing*; Yang, Bei*; Wang, Xiao; Wei, Jia; Lu, Zongyang; Zhang, Yuxi; Wu, Jing; Huang, Xingxu; Yang, Li; Chen, Jia.Base editing with a Cpf1-cytidine deaminase fusion.NATURE BIOTECHNOLOGY. Apr 2018. 36(4):324-327.
- 33. Lei, Liqun*; Chen, Hongquan*; Xue, Wei*; Yang, Bei*; Hu, Bian*; Wei, Jia; Wang, Lijie; Cui, Yiqiang; Li, Wei; Wang, Jianying; Yan, Lei; Shang, Wanjing; Gao, Jimin; Sha, Jiahao; Zhuang, Min; Huang, Xingxu; Shen, Bin; Yang, Li; Chen, Jia.APOBEC3 induces mutations during repair of CRISPR-Cas9-generated DNA breaks.NATURE STRUCTURAL & MOLECULAR BIOLOGY. Jan 2018. 25(1):45-52.
- 34. Wang, Lijie*; Xue, Wei*; Yan, Lei*; Li, Xiaosa; Wei, Jia; Chen, Miaomiao; Wu, Jing; Yang, Bei; Yang, Li; Chen, Jia.Enhanced base editing by co-expression of free uracil DNA glycosylase inhibitor.CELL RESEARCH. Oct 2017. 27(10):1289-1292.
- 35. Yang, Bei*; Li, Xiaosa; Lei, Liqun; Chen, Jia.APOBEC: From mutator to editor.JOURNAL OF GENETICS AND GENOMICS. Sep 2017. 44(9):423-437.
项目
-
1. 国家自然科学基金青年项目,国家自然科学基金委,项目负责人
-
2. 2021-国家自然科学基金面上项目,国家自然科学基金委,项目负责人
-
3. 国家重点研发计划“谱系或代谢示踪新标记物的鉴定”,中华人民共和国科技部,课题参与人员(非负责人)
-
4. 国家科技重大专项‘艾滋病疫苗研究’,中华人民共和国科技部,课题参与人员(非负责人)
-
5. 2023-上海市2023年度“科技创新⾏动计划” ⾯上项⽬,上海市科学技术委员会,项目负责人
-
6. 国家科技重大专项”新型基因编辑技术在癌症诊疗中的基础和转化研究“,中华人民共和国科技部,课题参与人员(非负责人)
-
7. 2024-国家自然科学基金面上项目,国家自然科学基金委,项目负责人
奖励
- 1. 2024,中国医药生物技术协会基因编辑技术分会委员
- 2. 2023,东方英才计划拔尖项目
- 3. 2023,上海生物化学与分子生物学学会-理事
- 4. 2020,中国性病艾滋病防治协会,艾滋病病毒学专业委员会委员
- 5. 2020、2021、2022、2023、2024,上海科技大学优秀教师
- 6. 2016,浦江人才计划
课题组成员及合影
-
姓名:戚琪莲
身份:工程师
在组时间:2016/01-至今
邮箱:qiql@@shanghaitech.edu.cn
-
姓名:张仙芳
身份:工程师
在组时间:2016/07-至今
邮箱:zhangxf1@@shanghaitech.edu.cn
-
姓名:王琪
身份:博士研究生
在组时间:2020-至今
邮箱:wangqi3@@shanghaitech.edu.cn
-
姓名:罗雅心
身份:博士研究生
在组时间:2021-至今
邮箱:luoyx1@@shanghaitech.edu.cn
-
姓名:候耀峰
身份:博士研究生
在组时间:2022-至今
邮箱:houyf2022@@shanghaitech.edu.cn
-
姓名:王强
身份:硕士研究生
在组时间:2023-至今
邮箱:wangqiang2023@@shanghaitech.edu.cn
-
姓名:陈佳妍
身份:硕士研究生
在组时间:2023-至今
邮箱:chenjy22023@@shanghaitech.edu.cn
-
姓名:何筱梦
身份:硕士研究生
在组时间:2024-至今
邮箱:hexm2024@@shanghaitech.edu.cn
-
姓名:尚欣迪
身份:硕士研究生
在组时间:2024-至今
邮箱:shangxd2024@@shanghaitech.edu.cn
-
姓名:陈晓湉
身份:硕士研究生
在组时间:2024-至今
邮箱:chenxt2024@@shanghaitech.edu.cn
|

















