课题组长
姓名:
张力烨副教授 研究员 博士生导师, PhD, 副教授
职务:
所在院所:
生命科学与技术学院
荣誉称号:
教育经历:
-
2003/09—2007/07,上海交通大学,学士
-
2007/08—2012/05,美国宾夕法尼亚州立大学,博士
博士后及工作经历:
- 2012/06—2017/05,美国波士顿大学医学院,博士后
- 2017/05—2025/09, 上海科技大学,生命科学与技术学院,助理教授(TENURE-TRACK)
- 2025/09—至今,上海科技大学,生命科学与技术学院,副教授(Tenured)
课题组简介
研究内容:
研究组侧重于使用多组学大数据分析,并整合单细胞测序,并建立新的计算方法来来研究肝癌发生,转移的过程中的遗传以及表观遗传发病机制。其中关注以下几点: 1. 研究癌症发生,转移的过程中基因组进化规律。 2. 开发新的针对单细胞组学的计算方法来研究癌症的免疫微环境 3. 通过多组学数据整合分析以及开发新的生物信息学算法来理解复杂生物学问题,过程和机制
研究成果展示
1.生物学方面: 1.1 2024年课题组和复旦中山医院合作研究肝癌转移过程中,转移过程的克隆演化规律,工作发表于2024年Cancer Cell,链接为https://doi.org/10.1016/j.ccell.2023.11.010。 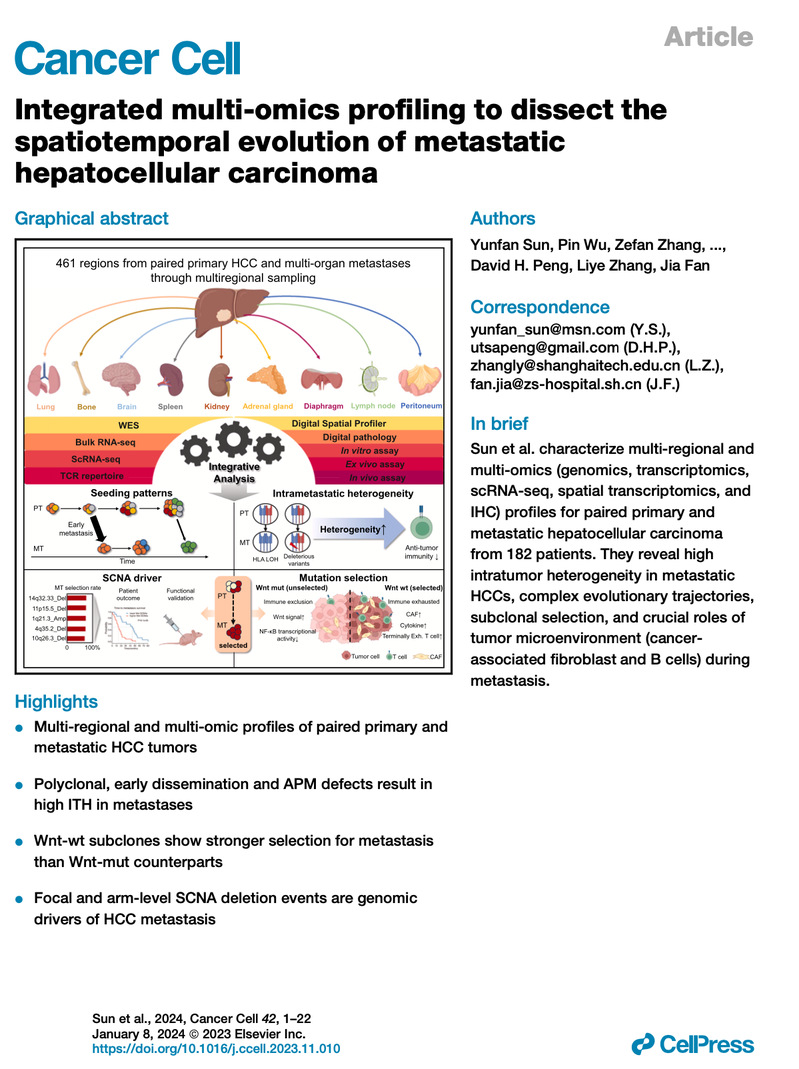
1.2. 课题组与合作者基于TGFbeta诱导EMT细胞模型模拟转移过程中表观遗传重编程,发现过程中染色体3D结构没有发生变化只是由于TF介导的增强子激活和关闭所调控,文章发表于2020年Molecular Therapy 链接:https://doi.org/10.1016/j.ymthe.2020.05.026。 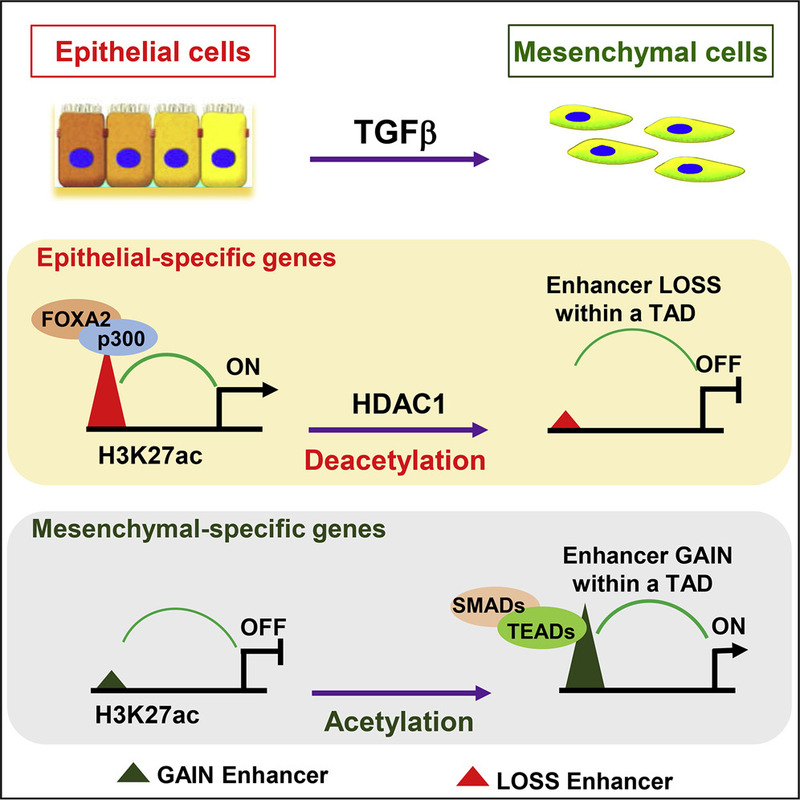
1.3 基于酵母的进化实验,我们发现酵母进化过程中多步进化的规律,以及通过计算大数据分析针对基因缺失使用同源基因突变来进行适应的普遍规律,文章发表于2023年JGG杂志,文章链接:https://doi.org/10.1016/j.jgg.2023.02.012。
2. 方法学方面: 3.1 实验室开发了针对scATAC-Seq的方法整合分析方法epiConv,传统方法是基于针对scRNA-Seq方法来整合scATAC-Seq,工作发表于2022年BMC Bioinformatics,文章链接:https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-022-04858-w 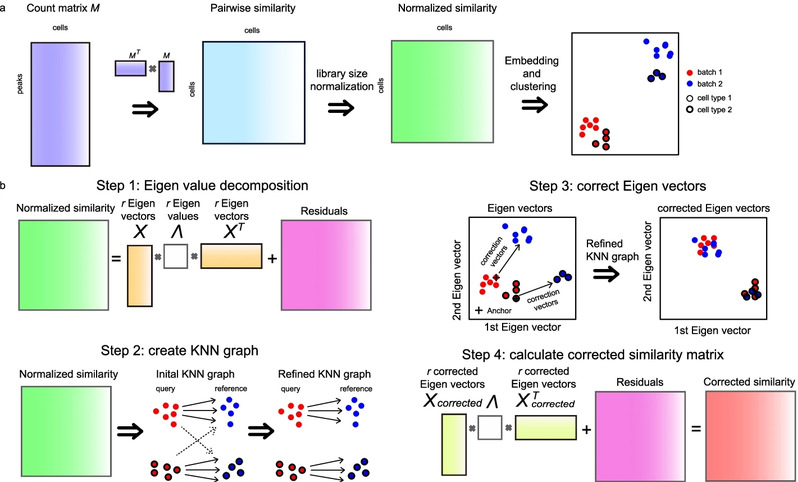
3.2 实验室开发了针对scRNA-Seq中高度稳定基因的标准化方法ISnorm,可以更好处理转录组整体量变化的场景,工作发表于2020年NAR Genomics and Bioinformatics 文章链接:https://doi.org/10.1093/nargab/lqaa059。
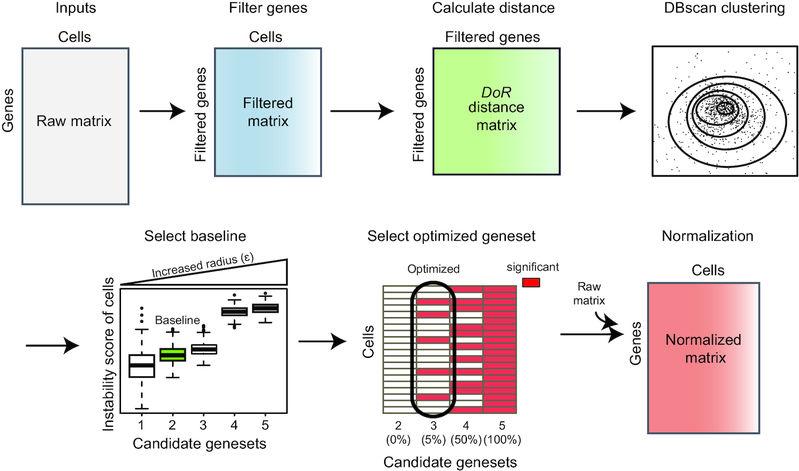
3.3 实验室开发了一个针对临床低深度测序样本构建病人多肿瘤样本构建准确系统进化树的新算法(PTI)。文章题目为“Phylogenetic Tree Inference: A Top-Down Approach to Track Tumor Evolution”,发表于2020年的Frontiers in Genetics杂志。
工具见实验室Github网页:https://github.com/bioliyezhang
代表性论文(*第一作者,#通讯作者)
- 1. Zhang, Liye#*; Yang, Chih-Sheng; Varelas, Xaralabos; Monti, Stefano#; Zhang, Liye#*; Yang, Chih-Sheng; Varelas, Xaralabos; Monti, Stefano#.Altered RNA editing in 3 ' UTR perturbs microRNA-mediated regulation of oncogenes and tumor-suppressors.SCIENTIFIC REPORTS. 2016. 6.
- 2. Voena, Claudia*; Varesio, Lydia M.; Zhang, Liye; Menotti, Matteo; Poggio, Teresa; Panizza, Elena; Wang, Qi; Minero, Valerio G.; Fagoonee, Sharmila; Compagno, Mara; Altruda, Fiorella; Monti, Stefano; Chiarle, Roberto#.Oncogenic ALK regulates EMT in non-small cell lung carcinoma through repression of the epithelial splicing regulatory protein 1.ONCOTARGET. 2016. 7(22):33316-33330.
- 3. Chapuy, Bjoern*; Roemer, Margaretha G. M.; Stewart, Chip; Tan, Yuxiang; Abo, Ryan P.; Zhang, Liye; Dunford, Andrew J.; Meredith, David M.; Thorner, Aaron R.; Jordanova, Ekaterina S.; Liu, Gang; Feuerhake, Friedrich; Ducar, Matthew D.; Illerhaus, Gerald; Gusenleitner, Daniel; Linden, Erica A.; Sun, Heather H.; Homer, Heather; Aono, Miyuki; Pinkus, Geraldine S.; Ligon, Azra H.; Ligon, Keith L.; Ferry, Judith A.; Freeman, Gordon J.; van Hummelen, Paul; Golub, Todd R.; Getz, Gad; Rodig, Scott J.; de Jong, Daphne; Monti, Stefano; Shipp, Margaret A.#.Targetable genetic features of primary testicular and primary central nervous system lymphomas.BLOOD. 2016. 127(7):869-881.
- 4. Chapuy, Bjoern*; Cheng, Hongwei; Watahiki, Akira; Ducar, Matthew D.; Tan, Yuxiang; Chen, Linfeng; Roemer, Margaretha G. M.; Ouyang, Jing; Christie, Amanda L.; Zhang, Liye; Gusenleitner, Daniel; Abo, Ryan P.; Farinha, Pedro; von Bonin, Frederike; Thorner, Aaron R.; Sun, Heather H.; Gascoyne, Randy D.; Pinkus, Geraldine S.; van Hummelen, Paul; Wulf, Gerald G.; Aster, Jon C.; Weinstock, David M.; Monti, Stefano; Rodig, Scott J.; Wang, Yuzhuo; Shipp, Margaret A.#.Diffuse large B-cell lymphoma patient-derived xenograft models capture the molecular and biological heterogeneity of the disease.BLOOD. 2016. 127(18):2203-2213.
- 5. Hiemer, Samantha E.*; Zhang, Liye; Kartha, Vinay K.; Packer, Trevor S.; Almershed, Munirah; Noonan, Vikki; Kukuruzinska, Maria; Bais, Manish V.; Monti, Stefano; Varelas, Xaralabos#.A YAP/TAZ-Regulated Molecular Signature Is Associated with Oral Squamous Cell Carcinoma.MOLECULAR CANCER RESEARCH. 2015. 13(6):957-968.
- 6. Suematsu, Takuma*; Zhang, Liye; Aphasizheva, Inna; Monti, Stefano; Huang, Lan; Wang, Qi; Costello, Catherine E.; Aphasizhev, Ruslan#.Antisense Transcripts Delimit Exonucleolytic Activity of the Mitochondrial 3 ' Processome to Generate Guide RNAs.MOLECULAR CELL. 2016. 61(3):364-378.
- 7. Aphasizheva, Inna*; Zhang, Liye; Wang, Xiaorong; Kaake, Robyn M.; Huang, Lan; Monti, Stefano; Aphasizhev, Ruslan#.RNA Binding and Core Complexes Constitute the U-Insertion/Deletion Editosome.MOLECULAR AND CELLULAR BIOLOGY. 2014. 34(23):4329-4342.
- 8. Zhang, Liye*; Ma, Hong#; Pugh, B. Franklin#.Stable and dynamic nucleosome states during a meiotic developmental process.GENOME RESEARCH. 2011. 21(6):875-884.
- 9. Rhee, Ho Sung*; Bataille, Alain R.; Zhang, Liye; Pugh, B. Franklin#.Subnucleosomal Structures and Nucleosome Asymmetry across a Genome.CELL. 2014. 159(6):1377-1388.
- 10. Zhang, Liye*; Sement, Francois M.; Suematsu, Takuma; Yu, Tian; Monti, Stefano; Huang, Lan; Aphasizhev, Ruslan; Aphasizheva, Inna#.PPR polyadenylation factor defines mitochondrial mRNA identity and stability in trypanosomes.EMBO JOURNAL. 15 Aug 2017. 36(16):2435-2454.
- 11. Wu, Pin*; Hou, Linjun; Zhang, Yingdong; Zhang, Liye#.Phylogenetic Tree Inference: A Top-Down Approach to Track Tumor Evolution.FRONTIERS IN GENETICS. 07 Feb 2020. 10.
项目
-
1. 癌症中研究RNA编辑调节蛋白表达的机制,国家自然科学基金,项目负责人
-
2. 自发性早产的动因学研究,科技部国家重点研发项目,课题参与人员(非负责人)
课题组成员及合影
-
姓名:赵晓静
身份:工程师
在组时间:2017/08-至今
邮箱:zhaoxj@shanghaitech.edu.cn
-
姓名:王巩
身份:博士研究生
在组时间:2022/11-至今
邮箱:wanggong2022@shanghaitech.edu.cn
-
姓名:娄力木
身份:博士研究生
在组时间:2022/11-至今
邮箱:loulm2022@shanghaitech.edu.cn
-
姓名:王宇坤
身份:博士研究生
在组时间:2023/05-至今
邮箱:wangyk2022@shanghaitech.edu.cn
-
姓名:马帅
身份:博士研究生
在组时间:2023/09-至今
邮箱:mashuai2023@shanghaitech.edu.cn
-
姓名:王永通
身份:博士研究生
在组时间:2024/03-至今
邮箱:wangyt22023@shanghaitech.edu.cn
-
姓名:张欣欣
身份:硕士研究生
在组时间:2023/11-至今
邮箱:zhangxx2023@shanghaitech.edu.cn
-
姓名:苏莉雅
身份:硕士研究生
在组时间:2023/09-至今
邮箱:suly2023@shanghaitech.edu.cn
-
姓名:汪恒
身份:硕士研究生
在组时间:2023/09-至今
邮箱:wangheng12023@shanghaitech.edu.cn
-
姓名:卓言
身份:硕士研究生
在组时间:2022/06-至今
邮箱:zhuoyan2025@shanghaitech.edu.cn
-
姓名:郑坤
身份:本科生
在组时间:2025/09-至今
邮箱:zhengkun2023@shanghaitech.edu.cn
-
姓名:高铭
身份:访问学生
在组时间:2025/09-至今
邮箱:gaoming2021@sjtu.edu.cn
|




















